Por: Mariana Mota, DCEB - Horticultura/CEER, Instituto Superior de Agronomia
MECANISMO DA INCOMPATIBILIDADE GAMETOFÍTICA
As angiospérmicas são o grupo de plantas mais evoluído e o seu sucesso resulta em grande parte da promoção da polinização cruzada como estratégia reprodutiva. O sistema de incompatibilidade gametofítica (GSI) representa uma destas estratégias, ocorrendo em várias famílias, nomeadamente nas rosáceas (onde se encontram as prunóideas e pomóideas).
A expressão deste sistema leva a que a autopolinização não resulte numa produção viável e rendível de fruto, fazendo com que o vingamento dependa da polinização cruzada. A autoincompatibilidade gametofítica da família Rosaceae é controlada por um único locus, o chamado S-locus ou Sterility-locus e é determinada pelas formas desse locus. Resumidamente, o S-locus contém 2 genes: um gene que codifica uma S-RNase (S de S-locus e RNase de ribonuclease) expressa extracelularmente no pistilo e o gene SFB (de S-locus F-Box; também designado por alguns grupos de investigação por SLF) que codifica uma proteína F-Box expressa no pólen. A composição do S-locus, definida pela combinação complementar do alelo S da S-RNase e do SFB é designada por S-haplótipo.
Na GSI, a incompatibilidade é determinada por uma constituição haplóide do pólen e diplóide do pistilo, estabelecendo-se a interacção durante o crescimento do tubo polínico no estilete, onde se produz a inibição do desenvolvimento do tubo polínico. Assim, o pólen pode germinar e penetrar o estigma, mas o tubo polínico não pode crescer completamente através do estilete, se o alelo S do grão de pólen é igual a um dos alelos presentes nas células do estilete (Fig. 1), o que acontece sempre que ocorre autopolinização.
Existem três níveis de compatibilidade entre cultivares com GSI. Se duas cultivares diferentes compartilharem todos os S haplótipos, elas serão incompatíveis; se elas diferirem em todos os S-loci serão totalmente compatíveis e se elas partilharem um dos S-loci elas serão semi-compatíveis (Fig. 1).
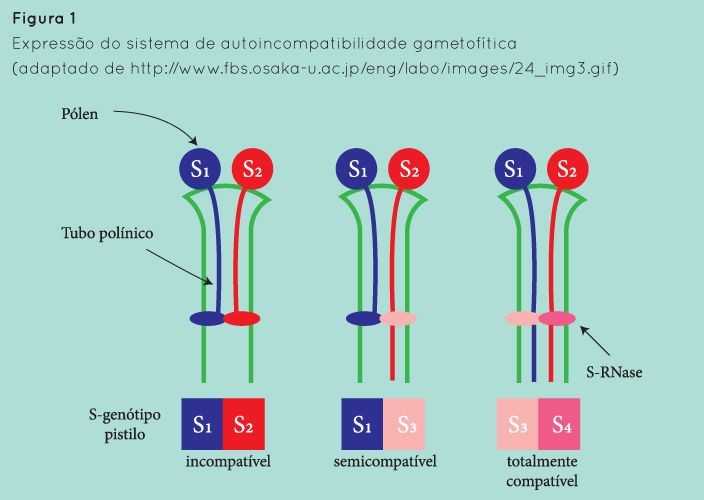
A expressão dos dois alelos é codominante, estando os alelos S das células do estilete ambos envolvidos na influência exercida no crescimento do tubo polínico e controlando este locus de incompatibilidade tanto a autoincompatibilidade como a incompatibilidade cruzada intra-específica. Estes alelos codificam a produção das S-RNases que ocorre na matriz intracelular do tecido transmissor. Um modelo de degradação da proteína foi anteriormente proposto para explicar a rejeição específica do tubo polínico pelas S-RNases nas rosáceas: segundo este, a S-FBox reconhece especificamente a RNase diferente do seu S-haplótipo e medeia a sua degradação através de um proteassoma (via ubiquitina -proteassoma -26S), num processo que envolve a participação de diferentes genes, como é o caso do gene da transglutaminase. Investigações recentes apontam para que este modelo se adeque mais às pomóideas, havendo ligeiras variações a este comportamento na expressão da GSI nas prunóideas.
DETERMINANTES NO PISTILO
Tal como noutras espécies, as S-RNases são glicoproteínas consideradas responsáveis pela expressão do mecanismo de GSI no pistilo, tanto nas pomóideas como nas prunóideas. É-lhes atribuído um papel citotóxico que, em cruzamentos incompatíveis, é responsável pela rejeição do pólen na parte distal do estilete. Nas rosáceas, estas proteínas encontram-se organizadas em 5 regiões conservadas, tendo apenas uma região hipervariável. As regiões conservadas são importantes para a especificidade ribonucleásica, enquanto a região hipervariável parece estar mais envolvida no reconhecimento. Tanto nas pomóideas como nas prunóideas, existe um intrão na região hipervariável e, nas prunóideas, existe um intrão adicional após o sinal peptídico, no início da proteína madura. A presença dos intrões na estrutura do gene da S-RNase é fundamental para a sua identificação expedita a nível molecular por PCR (polymerase chain reaction, reacção em cadeia da polimerase), aspecto que se detalhará mais na parte II deste artigo.
DETERMINANTES NO PÓLEN
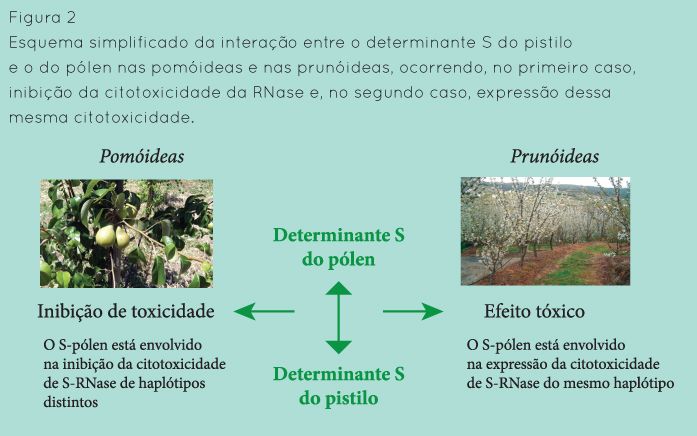
O determinante do S-locus no pólen foi estudado primeiro nas prunóideas. Enquanto nas prunóideas apenas existe um gene F-Box no pólen que determina a expressão do mecanismo, nas pomóideas existem múltipos genes F-Box (designados por SFBBs, de S-locus F-Box Brothers), responsáveis pela especificidade do mecanismo. Todos estes genes mostram polimorfismos específicos do S-haplótipo, ligação à RNase e expressão específica no pólen. Esta organização divergente nas pomóideas e prunóideas levou a que se estudasse mais aprofundadamente o tema. Os genes S do pólen têm um domínio F-Box, duas zonas variáveis e duas zonas hipervariáveis. As zonas variáveis e hipervariáveis estão envolvidas no reconhecimento. As proteínas F-Box são consideradas receptores, capazes de incorporar proteínas dirigidas para degradação proteassómica (via proteolítica mediada pela ubiquitina). Estudos recentes revelaram que estas proteínas interagem especificamente com as S-RNases não próprias (não do mesmo haplótipo), inibindo a sua actividade ribonucleásica e citotóxica dentro do tubo polínico nos cruzamentos compatíveis (no caso das pomóideas) ou interagindo com a S-RNase própria para a deixar activa no caso de cruzamentos incompatíveis, bloqueando o desenvolvimento dos tubos polínicos (no caso das prunóideas) (Fig. 2). Nas pomóideas, é preciso ainda esclarecer o papel dos múltiplos SFBB genes e como múltiplas proteínas F-Box conseguem reconhecer um grande grupo de S-RNases não próprias, mas pensa-se que o mecanismo varia. Em Petunia (solanácea) verificou-se que três tipos divergentes de S- haplótipos no pólen conseguiam reconhecer cada um um conjunto de S-RNases não próprias, no que foi chamado um reconhecimento colaborativo não próprio. Este modelo propõe que as múltiplas proteínas F-Box são necessárias no seu conjunto para que todas as S-RNases não próprias sejam colectivamente reconhecidas e a sua capacidade citotóxica inibida. Este modelo poderia aplicar-se às pomóideas. Neste caso, aparentemente não há interacção entre nenhum dos genes SFBs do S-locus e a S-RNase própria, permitindo que a S-RNase fique activa e impeça a fertilização. Na polinização cruzada, as proteínas SFB/SLF interagem com as S-RNases não próprias, impedindo a sua citotoxicidade e permitindo a fertilização.
MUTANTES E QUEBRA DA AUTOINCOMPATIBILIDADE: PRUNÓIDEAS VS POMÓIDEAS
A diferença entre pomóideas e prunóideas tem consequências ao nível do comportamento dos mutantes associados à quebra da autoincompatibilidade. Nas prunóideas, já foram identificados vários mutantes em que inserções ou deleções nos genes S do pólen resultam na quebra da GSI, sugerindo que, de facto, o determinante S do pólen actua protegendo a sua própria S-RNase de um inibidor geral, permitindo que exerça a sua função citotóxica. Quando esse gene se encontra mutado, a acção protectora não se verifica, a S-RNase é degradada e deixa de exercer a sua acção tóxica. Pensa-se que, neste grupo, a disfunção dos determinantes S do pistilo ou do pólen leva à autocompatibilidade e que a autocompatibilidade é conferida até por baixos níveis de transcrição de genes codificadores das S-RNases, que levam a baixos níveis de acumulação de S-RNases no estilete. Nas pomóideas ainda não foram identificados mutantes não funcionais no pólen, ligados à autocompatibilidade, tal como não foi identificado nenhum mecanismo de inibição da acção tóxica da S-RNase independente do gene S do pólen. De acordo com o modelo proposto, uma mutação no determinante S do pólen não confere autocompatibilidade mas sim impede a fertilização pelo pólen mutado. Neste grupo, associou-se a quebra da GSI a mutações no gene S no pistilo, tendo sido já identificadas mutações na sequência genética mas também alterações ao nível do mecanismo de transcrição que poderão estar relacionadas com a disfunção do gene S no pistilo. Nestas, disfunções a nível do determinante S do pólen resultam em pólen incompatível tanto em autopolinização como em polinização cruzada.
É, no entanto, de notar que há já várias referências a outros genes não localizados no S-locus e não directamente envolvidos na determinação deste mecanismo S como estando envolvidos na GSI baseada na S-RNase. É o caso do gene da transglutaminase, ou o CUL 1, entre outros. Assim, mutações de genes externos ao S-locus contribuem também para a quebra da autoincompatibilidade.
APLICAÇÕES EM HORTICULTURA
A aplicação mais disseminada é a identificação por via molecular dos conteúdos alélicos dos diferentes haplótipos. A clonagem dos genes S-RNase e SFB permitiu a obtenção a nível molecular de S-haplótipos [parte II], os quais foram fundamentais para a determinação da compatibilidade cruzada em diversas fruteiras (Quadro 1). Esta abordagem tem permitido a definição de grupos de cultivares compatíveis entre si e a identificação de cultivares incompatíveis nas espécies em que a autoincompatibilidade é predominante. Tem permitido também já a identificação de alelos mutados que ocasionam por vezes genótipos autoférteis, muito úteis do ponto de vista agronómico mas também em termos de inclusão em programas de melhoramento. A identificação molecular dos S-haplótipos tem também aplicação na selecção assistida por marcadores, permitindo a identificação precoce de indivíduos autocompatíveis.
Uma outra aplicação interessante prende-se com a possibilidade de obter cultivares autoférteis por transformação genética. Mutantes obtidos por silenciamento da S-RNase estão já disponíveis e revelam que esta estratégia pode constituir uma ferramenta sólida para o melhoramento.
Bibliografia
- Dondini, L.; Franceschi, P.; Sansavini, S. (2013) - Incompatibility in fruit tree species: S-Locus structure and molecular mechanism of pistil and pollen determinants. Acta Horticulturae 981: 31-40;
- Franceschi, P. de; Dondini, L.; Sanzol, J. (2012) - Molecular bases and evolutionary dynamics of self-incompatibility in the Pyrinae (Rosaceae). Journal of Experimental Botany 63: 4015-4032;
- Goldway, M.; Sapir, G.; Stern, R. A.; (2007). Molecular basis and Horticultural application of the gametophytic self-incompatibility system in rosaceous tree fruits. Plant Breeding Reviews 28: 215 – 237;
- Mota, M.; Tavares, L.; Oliveira, CM. (2007) - Identification of S-alleles in pear (Pyrus communis L.) cv. ‘Rocha’ and other European cultivars. Scientia Horticulturae 113:13-19
- Nettancourt, D. (1977) - Incompatibility in Angiosperms. Ed. Springer – Verlag, Berlin;
- Sanzol, J. (2009) - Pistil-function breakdown in a new S-allele of european pear, S21 degrees , confers self-compatibility. Plant Cell Reports 28: 457-467;
- Sassa, H.; Kakui, H.; Minamikawa, M. (2010) - Pollen-expressed F-box gene family and mechanism of S-RNase-based gametophytic self-incompatibility (GSI) in Rosaceae. Sexual Plant Reproduction 23: 39-43;
- Sassa, H.; Kakui, H.; Miyamoto, M.; Suzuki, Y.; Hanada, T.; Ushijima, K.; Kusaba, M.; Hirano, H.; Koba, T. (2007) - S locus F-box brothers: multiple and pollen-specific F-box genes with S haplotype-specific polymorphisms in apple and Japanese pear. Genetics 175: 1869-1881.
- Tao, R.; Matsumoto, D. (2012) - S locus mutation and self-compatibility in stone fruits. Acta Horticulturae 967: 105-111.